Genomische Selektion
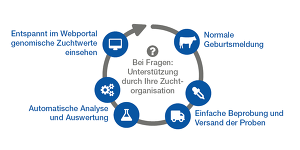
Während noch bis 2010 die gesamte Selektion von Jungtieren über den Pedigree-Index (also auf Basis von Eltern-Zuchtwerten) erfolgen musste, bieten die genomischen Zuchtwerte heute einen gezielteren Einblick in das genetische Potential der nächsten Generation. Um genomische Zuchtwerte Schätzen zu können, muss zunächst im Labor eine (Geno-)Typisierung erfolgen.
Bei der Typisierung wird die DNA eines Lebewesens auf eine Vielzahl ausgewählter Marker hin untersucht. Diese Marker werden SNP (Single Nucleotide Polymorphism) genannt. SNP sind vererbbare Punktmutationen und treten daher in Form einer ausgetauschten Base im DNA-Strang auf. In der Regel werden ca. 50.000 SNP gleichzeitig im Labor untersucht. Die Laborergebnisse können dabei für jeden SNP in drei Kombinationen auftreten (zweimal das Allel 1 (homozygot), einmal Allel 1 und einmal Allel 2 (heterozygot) oder zweimal Allel 2 (homozygot)). Selten liegt ein SNP direkt innerhalb eines Gens und bildet z.B. die kausale Mutation für unterschiedliche Merkmalsausprägungen (Phänotypen). Dennoch haben SNPs einen hohen Informationsgehalt, wenn sie in der Nähe wichtiger Gene liegen.

Schon wenige Tage nach der Geburt wird bei Kälbern eine Ohrstanzprobe gezogen und zur Typisierung ins Labor geschickt (Foto: D. Warder)
Soll ein Tier typisiert werden, schickt der Landwirt eine Probe mit genetischem Material (z. B. Blut, Gewebe oder Haare) und einem Untersuchungsantrag an ein akkreditiertes Labor. Dort wird die DNA aus der Probe extrahiert. Anschließend wird die DNA vervielfältigt und fragmentiert. Die DNA-Fragmente können auf Veränderungen (SNPs) untersucht werden. Dazu wird die fragmentierte DNA auf einen Chip aufgetragen, in dem quasi Andockstellen für die SNPs vorliegen, auf die die DNA untersucht werden soll. Je nachdem in welcher Ausprägung ein SNP vorliegt, verbindet sich der kurze Strang mit der passenden Andockstelle. Die SNP werden je nach Ausprägung mit einem Farbstoff (rot/grün) markiert. Über ein Lichtsignal, das bei der Anregung durch einen hochsensiblen Laserscanner von einem markierten SNP abgegeben wird, kann dann die Information über den SNP vom Computer ausgelesen werden. Da jeweils ein SNP von der Mutter und vom Vater vererbt wird, kann es drei mögliche Ausprägungen für eine Basenposition (Ort des SNP auf der DNA) geben. Daher gibt es entsprechend drei mögliche Farbsignale (rot/grün/rot und grün).
Die Ergebnisse der Typisierung übersendet das Labor anschließend an das Rechenzentrum in Verden (vit). Dort beginnt dann die Verarbeitung der Informationen und die Schätzung genomischer Zuchtwerte. Vom Einsenden der Probe bis zur Verarbeitung der Typisierungsergebnisse zu Zuchtwerten im vit vergehen ca. zwei bis drei Wochen.
Berücksichtigt werden in der Zuchtwertschätzung die SNP-Effekte. Je näher ein SNP einem Gen (mit Einfluss auf ein betrachtetes Merkmal) liegt, desto größer ist dessen Effekt im Zuchtwertschätzmodell auf das betrachtete Merkmal. Dies setzt natürlich voraus, dass das Gen und der SNP jeweils zusammen in den gleichen möglichen Ausprägungen auftreten. In diesem Fall spricht man von einer Kopplung. Die genomischen Zuchtwerte werden dann den Zuchtorganisationen und den Besitzern der Tiere über ein Onlineportal zur Verfügung gestellt.
Auf Basis der genomischen Zuchtwerte ist es dann Landwirten und Zuchtorganisationen möglich, gezielte Selektionsentscheidungen schon im ganz jungen Alter der Tiere zu treffen.
KuhVision und Herdentypisierung
In Deutschland ist die Typisierung von weiblichen Tieren der Rasse Deutsche Holsteins für alle Herdbuchbetriebe möglich. Grundsätzlich besteht die Möglichkeit, jederzeit die Typisierung einzelner Tiere zu beantragen. Es kann jedoch auch die gesamte weibliche Nachzucht nach der Geburt typisiert werden. Dazu schließen Betriebe einen Vertrag mit Ihrer Zuchtorganisation ab und erhalten dafür einen Preisnachlass bei den Typisierungskosten. Landwirte, die sich entschließen, Gesundheitsdaten zusätzlich zu erfassen, können im Projekt KuhVision einen weiteren Rabatt erhalten.
Mittlerweile sind über dieses System mehr als 18 % der Herdbuchkühe (> 234.000 Kühe) typisiert und liefern damit einen wertvollen Beitrag zur Verbesserung der genomischen Zuchtwertschätzung.
Bei einer Beteiligung an diesen Projekten ergeben sich für die Teilnehmer eine Vielzahl von Vorteilen:
- Genauer Überblick über den genetischen Status der Herde durch die Rücklieferung von genomischen Zuchtwerten mit hohen Sicherheiten
- Zuchtwerte für direkte Gesundheitsmerkmale als Hinweis auf die genetische Anfälligkeit von wirtschaftlich bedeutenden Krankheitskomplexen
- Verringerung der für die Remontierung der Herde benötigten Besamungen durch Auswahl der genetischen Spitze der Herde (eigene Schwerpunkte) für die Remonte
- Ermöglichung des sehr gezielten Einsatzes von gesextem Sperma, sodass sich der betriebliche Zuchtfortschritt erhöht
- Verkauf der für die eigene Remontierung nicht benötigten Nachzucht
- Aufzucht geringerer Anzahl Tiere notwendig (wenn die betriebliche Remontierungsrate dies erlaubt) - gezielterer Einsatz betrieblicher Ressourcen
- Gezielter Ausgleich genetischer Schwächen z.B. in der Tiergesundheit
- Durch Nutzung des BullenAnpaarungsProgramms (BAP) betriebsindividuelle Zuchtziele setzen und verfolgen
- Ermöglichung einer zielgerichteten Zucht auf genetisch hornlose Tiere durch Überblick über den Hornstatus
- Automatisierte Abstammungskontrolle der typisierten Tiere
- Vermeidung von Risikoanpaarungen für Erbfehler
- Verringerung / Begrenzung des Inzuchtgrades in der Herde
- Zuchtwertbenchmark innerhalb der am Projekt teilnehmenden Betrieben
Die Typisierung männlicher Kälber und Bullen erfolgt nur im Rahmen der Zuchtprogramme zur Selektion möglicher neuer Zuchtbullen und bei Deckbullen in Herdentypisierungsbetrieben.
Weitere Informationen finden Sie auf der Seite Richtig Züchten und auf den Seiten der deutschen Zuchtorganisationen.